11.2 Generazione di eventi distribuiti secondo una densità di probabilità fissata¶
Metodo hit and miss¶
Supponiamo di avere la seguente distribuzione di probabilità: $$P(x) = \begin{cases} 0.6 \quad x<0.5\\ 1.7 \quad x>0.5 \end{cases}$$
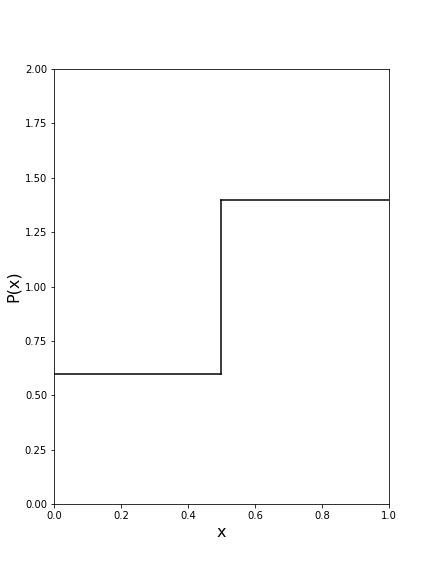
e di voler generare valori di $x$ distribuiti secondo $P(x)$
Procediamo nel modo seguente:
- Troviamo un numero $Y$ tale che $P(x) < Y, \,\forall x$. Nel nostro caso potremmo prendere $Y = 2$, per esempio.
- Generiamo un numero casuale $x_0$ in [0,1], in modo uniforme.
- Generiamo un secondo numero casuale $y_0$ in [0,$Y$]
- Teniamo il numero $x_0$ nel sample se $y_0 < P(x_0)$ altrimenti lo scartiamo e generiamo un nuovo valore $x_0$.
La probabilità di tenere un punto nel bin di sinistra è: $$P_{sx}=P(x < 0.5)\cdot P(y < 0.6) = 0.5\cdot\frac{0.6}{2}$$ La probabilità di tenere un punto nel bin di destra è: $$P_{dx}=P(x > 0.5)\cdot P(y < 1.7) = 0.5\cdot\frac{1.4}{2}$$ Quindi il rapporto $R$ fra le due probabilità, ovvero il rapporto fra il numero di punti accettati in $x < 0.5$ e il numero di punti accettati in $x > 0.5$ è, come deve: $$ R = \frac{P_{sx}}{P_{dx}} = \frac{0.6}{1.7}$$